Exemple
La fenêtre suivante contient les séquences de trois allèles différents (A, B et O) du gène
des groupes sanguins. La séquence est colorée par type de nucléotide (
Colour > Nucleotide)
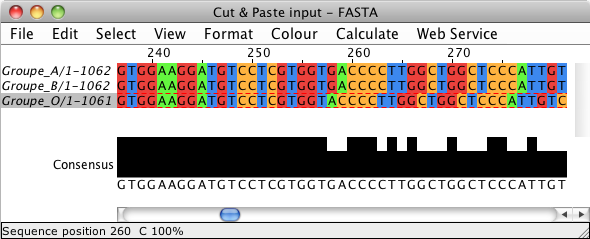
On peut repérer un décalage d'un nucleotide dû à une délétion en position 258 pour la séquence "O".
Un clic droit + Maj sur l'adénine placée en position 258 permet de la décaler en position 259.
Exemple

Le résultat s'affiche dans une fenêtre de calcul et les alignements peuvent ensuite être importés dans
une nouvelle fenêtre de visualisation en cliquant sur le bouton "View in alignment editor".
Remarque 1 :Notez qu'en dessous de chaque paire de séquences alignées est indiqué le Pourcentage d'identité.
Par exemple pour ces trois séquences les pourcentages sont :
On peut donc en déduire la matrice des distances suivante :
|
|
A
|
B
|
O
|
| A
|
0
|
0,38
|
0,09
|
| B
|
|
0
|
0,47
|
| O
|
|
|
0
|
Remarque 2 : Dans la fenêtre de calcul d'alignement, une barre verticale (|) indique une identité, un point (.)
dans un alignement de protéines indique une substitution par un acide aminé proche chimiquement (exemple valine/leucine),
une absence de caractère indique une différence.
Exemple
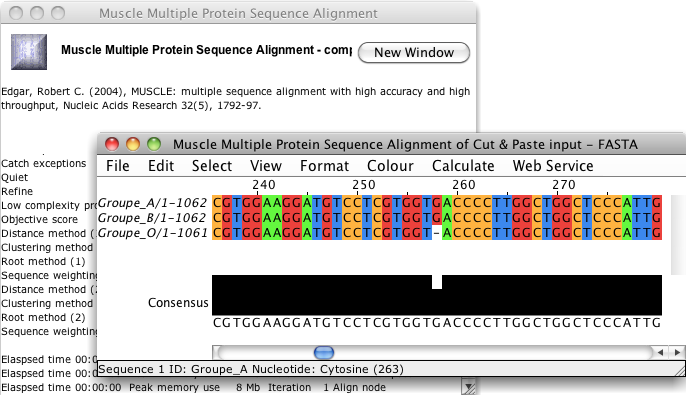
La fenêtre en arrière plan est la fenêtre d'information qui s'ouvre lorsque la tâche est lancée
et qui indique son déroulement jusqu'à exécution complète. La fenêtre au premier plan
correspond à la fenêtre de résultat. Pour des commodités de lecture, l'alignement a été
coloré par nucléotides et positionné au niveau de la délétion mise en évidence précédemment.
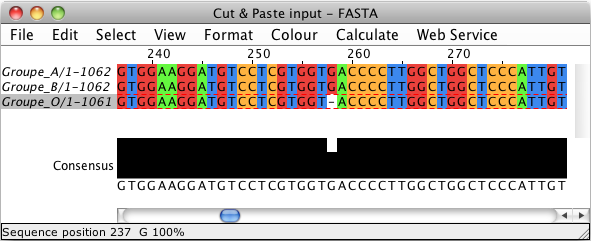
 On peut repérer un décalage d'un nucleotide dû à une délétion en position 258 pour la séquence "O".
Un clic droit + Maj sur l'adénine placée en position 258 permet de la décaler en position 259.
On peut repérer un décalage d'un nucleotide dû à une délétion en position 258 pour la séquence "O".
Un clic droit + Maj sur l'adénine placée en position 258 permet de la décaler en position 259.

 Le résultat s'affiche dans une fenêtre de calcul et les alignements peuvent ensuite être importés dans
une nouvelle fenêtre de visualisation en cliquant sur le bouton "View in alignment editor".
Le résultat s'affiche dans une fenêtre de calcul et les alignements peuvent ensuite être importés dans
une nouvelle fenêtre de visualisation en cliquant sur le bouton "View in alignment editor". La fenêtre en arrière plan est la fenêtre d'information qui s'ouvre lorsque la tâche est lancée
et qui indique son déroulement jusqu'à exécution complète. La fenêtre au premier plan
correspond à la fenêtre de résultat. Pour des commodités de lecture, l'alignement a été
coloré par nucléotides et positionné au niveau de la délétion mise en évidence précédemment.
La fenêtre en arrière plan est la fenêtre d'information qui s'ouvre lorsque la tâche est lancée
et qui indique son déroulement jusqu'à exécution complète. La fenêtre au premier plan
correspond à la fenêtre de résultat. Pour des commodités de lecture, l'alignement a été
coloré par nucléotides et positionné au niveau de la délétion mise en évidence précédemment.





