Exemple
La fenêtre suivante contient les séquences de trois allèles différents (A, B et O) du gène
des groupes sanguins.
La séquence sans coloration (par défaut).

La séquence est colorée par type de nucléotide.
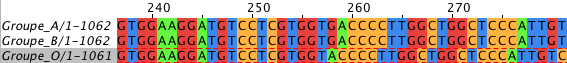
Remarque : on visualise ainsi un décalage entre séquences dû à une délétion en position 258 sur la séquence de l'allèle O.
Exemple
Les images suivantes représentent un même alignement de séquences de myoglobines de vertébrés, en utilisant des colorations différentes

Coloration BLOSUM62
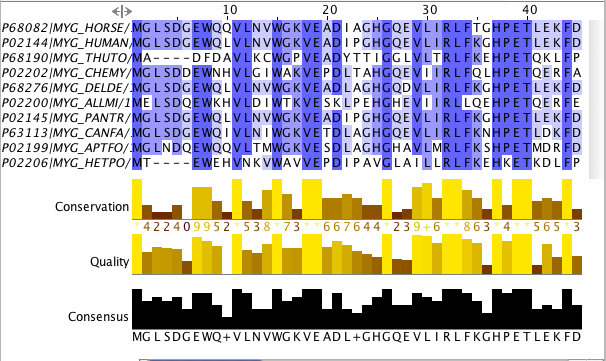
Coloration par pourcentage d'identité

Coloration par pourcentage d'identité avec l'option Coloration par degré de conservation.
Remarque : la discrimination entre les différents acides aminés est la plus forte avec ce type de coloration.
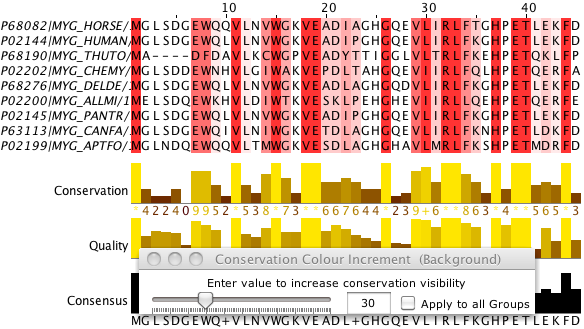
Coloration définie par l'utilisateur (tous les acides aminés sont colorés en rouge) avec l'option coloration par degré de conservation.
Pour télécharger le mode de coloration utilisé :
rouge.jc
 La séquence est colorée par type de nucléotide.
La séquence est colorée par type de nucléotide.  Remarque : on visualise ainsi un décalage entre séquences dû à une délétion en position 258 sur la séquence de l'allèle O.
Remarque : on visualise ainsi un décalage entre séquences dû à une délétion en position 258 sur la séquence de l'allèle O.
 Coloration BLOSUM62
Coloration BLOSUM62 Coloration par pourcentage d'identité
Coloration par pourcentage d'identité Coloration par pourcentage d'identité avec l'option Coloration par degré de conservation.
Coloration par pourcentage d'identité avec l'option Coloration par degré de conservation. Coloration définie par l'utilisateur (tous les acides aminés sont colorés en rouge) avec l'option coloration par degré de conservation.
Coloration définie par l'utilisateur (tous les acides aminés sont colorés en rouge) avec l'option coloration par degré de conservation.




