par Philippe Cosentino
Que ce soit pour des raisons de mesures sanitaires (élèves confinés à leur domicile), ou pour des raisons de contraintes techniques (incompatibilité logicielle, difficultés à installer certains logiciels), les logiciels hybrides (c’est à dire fonctionnant en ligne et hors ligne) ont la côte.
Dans un contexte d’enseignement distanciel, il est en effet peu réaliste d’imaginer que 35 élèves, équipés diversement en matériel informatique, pourront tous installer sur leur ordinateur personnel tel ou tel logiciel prévu pour tourner sur des machines sous Windows (certains étant d’ailleurs sur Mac).
Les applications « en ligne », quant à elles, ont l’avantage de tourner sur n’importe quelle machine, et même sur une tablette ou un simple téléphone.
C’est le cas du nouveau logiciel « Geniegen2 », qui, comme son nom l’indique, reprend le flambeau du logiciel gratuit Geniegen développé en son temps par Jean François Madre.
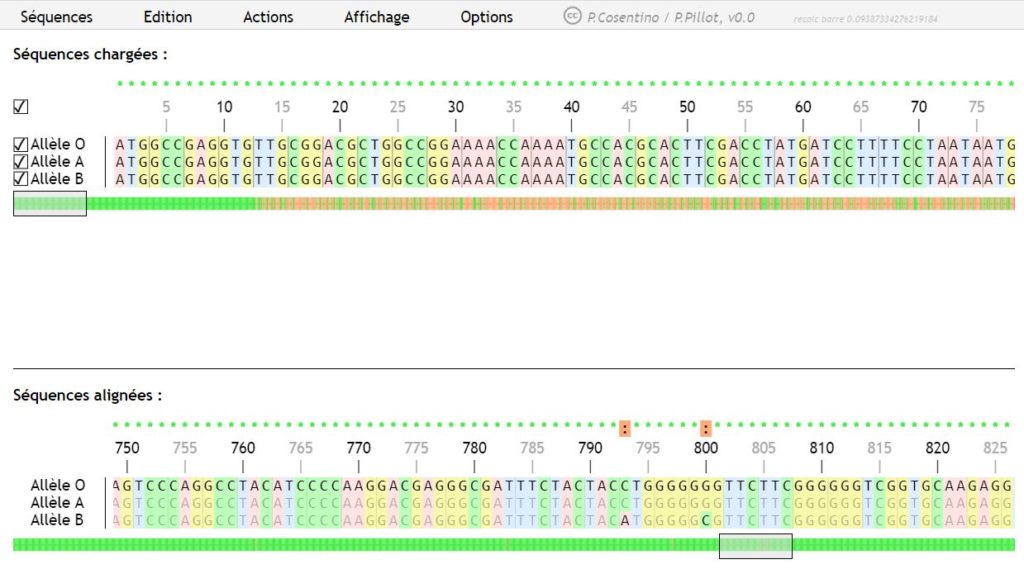
Ce logiciel permet, comme ses prédécesseur, de charger des séquences (par exemple au format .edi, le format d’Anagène), de les visualiser, de les aligner (afin de les comparer malgré les discontinuités) et de les traiter.
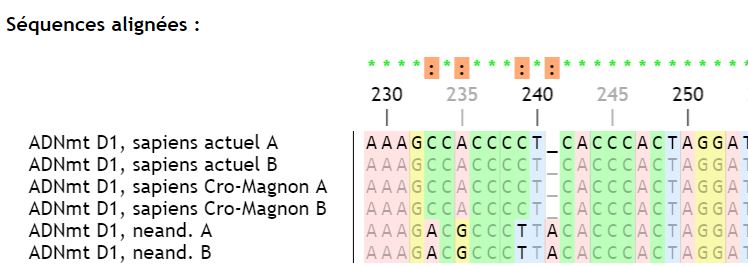
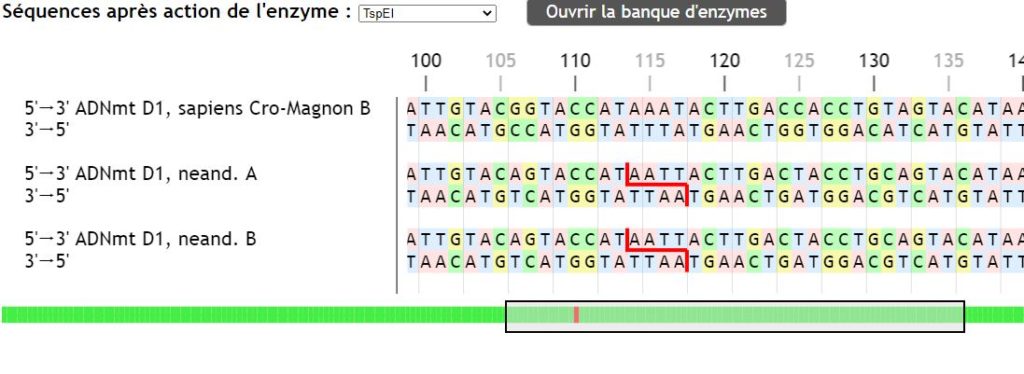
La visualisation des séquences est facilitée par un code de couleurs qui met en évidence les similarités et les différences. Ces dernières sont d’ailleurs directement identifiables (par des barres verticales rouges) dans l’ascenseur horizontal permettant de se déplacer dans la séquence.
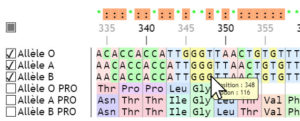
Les séquences polypeptidiques peuvent être affichées avec un code à 1 lettre par acide aminé, ou à 3 lettres (menu option). La numérotation peut aussi se faire par nucléotide pour par codon/résidu. On obtient par ailleurs la position exacte du curseur de la souris simplement en survolant la séquence.
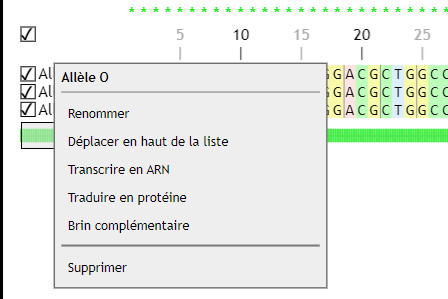
Des menus contextuels (clic-droit sur les titres des séquences) permettent des actions rapides, tandis qu’un système de coches permet des actions « groupées » sur plusieurs séquences.
L’alignement (menu « Actions/Aligner ») utilise un algorithme développé par Paul Pillot. Il permet de comparer des séquences présentant des discontinuités (délétions, insertions).
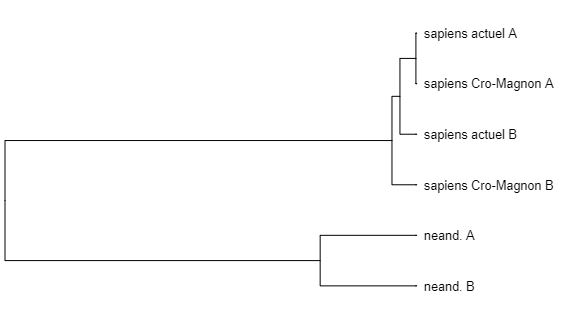
Il est également possible de construire un phénogramme (arbre) matérialisant les degrés de similitudes (et par hypothèse également de parenté) entre des séquences homologues. A noter : Geniegen2 accepte les fichiers au format .aln (Phylogène), et peut donc s’y substituer dans le cadre des ECE, si l’activité se limite à construire un tel arbre.
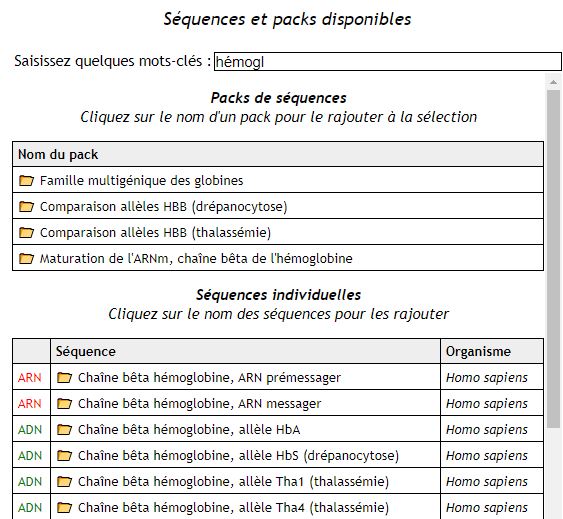
Geniegen2 dispose d’une banque interne de séquences, qui pour l’instant est rudimentaire, mais qui s’étoffera rapidement au fur et à mesure des demandes remontant des collègues (comme pour Mesurim2).
Les enzymes de restriction peuvent également être utilisées, l’enseignant peut choisir, dans une banque, une sélection d’enzymes, dont il pourra tester l’action sur les séquences sélectionnées.
Enfin, le logiciel laisse la possibilité à l’enseignant de créer un « lien direct » vers des séquences, à condition que celles-ci aient été prises dans la banque interne. L’avantage étant alors que l’élève n’a pas de séquences à récupérer ou à aller chercher, Geniegen2 s’ouvrant directement sur les séquences choisies par l’enseignant. Cette fonctionnalité devrait représenter une aide majeure pour les enseignants travaillant en distanciel ou selon un mode hybride.
Geniegen2 fonctionne sur Windows, Mac et Linux, mais également sur les tablettes Android (comme celles fournies par la région aux lycéens) et Apple (iPad), à condition d’avoir une connexion internet (pour l’instant), sur lesquelles les fonctionnalités tactiles (par exemple glisser la séquences de gauche à droite sans passer par l’ascenseur) sont prises en charge.
Lien
Application Geniegen2 en ligne