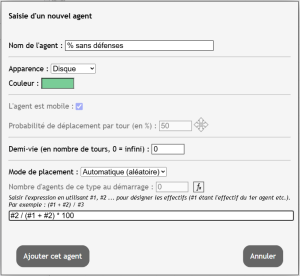
par Philippe Cosentino Cet article présente des fonctions avancées des effectifs dans le mode « expert » d’Edu’modèles multi-agents (algorithmique). Il ne s’adresse qu’aux utilisateurs déjà familiarisés avec ce logiciel.
Catégorie : Logiciels
Utiliser Tectoglob3D et le retard des ondes PmP pour mettre en évidence un épaississement de la croûte sous les Alpes
Travail original de Hubert Ferry et Jean-Luc Bérenguer, adapté par Jean-Luc Bérenguer et Philippe Cosentino. RÉSUMÉ En comparant, sur des sismogrammes, les temps d’arrivée des ondes sismiques, il est possible de mettre en évidence un épaississement crustal au niveau des Alpes.
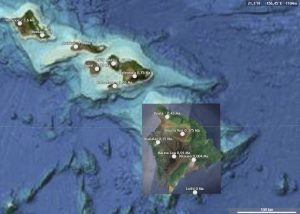
Etudier le volcanisme de point chaud (Hawaï) avec Tectoglob3D
par Philippe Cosentino RÉSUMÉ La mobilité des plaques tectoniques peut être étudiée à travers l’alignement des volcans de point chaud de l’archipel d’Hawaï. Plusieurs scenarii pédagogiques ont déjà été publiés sur ce sujet, mais presque tous reposent sur l’utilisation de Google Earth. Or la version « en ligne » de Google Earth ne permet pas de travailler …
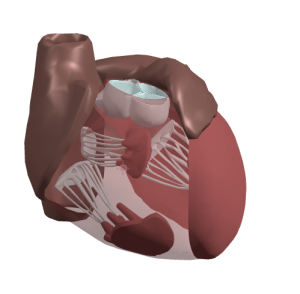
Oscar3D : un modèle anatomique numérique conçu pour l’enseignant de SVT
par Philippe Cosentino Lorsqu’on se représente une salle de SVT, la première image qui vient à l’esprit (après le microscope peut-être) est celle du squelette, ou de l’écorché. Si ce dernier reste un incontournable, il faut bien reconnaître qu’il a ses limites ; les organes sont souvent grossièrement modélisés, ou indissociables, il est difficile de …
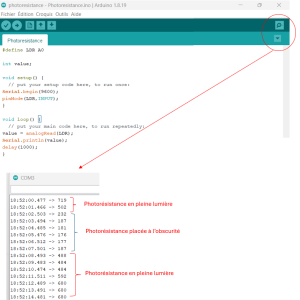
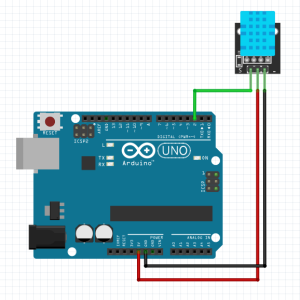
Mesurer la luminosité avec une photorésistance et une carte Arduino-TraAMs 2023/2024
RÉSUMÉ Dans le cadre du projet expérimental et numérique mené par les élèves de première enseignement scientifique , il est suggéré l’utilisation de capteurs associés à un microcontrôleur. Un groupe d’ élèves du lycée international de Valbonne a étudié à l’aide de photorésistances et de microcontrôleur ARDUINO la pollution lumineuse au sein de l’établissement. …
Mesurer l’humidité et la température du sol avec un capteur d’humidité et une carte Arduino
RÉSUMÉ Activité pour les secondes dans le cadre du thème 2 : « les enjeux contemporains de la planète – Vers une gestion durable des agrosystèmes ». Démarche expérimentale visant à rechercher et tester des solutions permettant de limiter la consommation d’eau pour l’arrosage des cultures.
Mesurer les variations de température dans l’eau d’une mare pédagogique avec un capteur de température et une carte Arduino -TraAMs 2023/2024
RÉSUMÉ Dans le cadre du projet expérimental et numérique mené par les élèves de première enseignement scientifique , il est suggéré l’utilisation de capteurs associés à un microcontrôleur. Un groupe d’élèves du lycée international de Valbonne a travaillé sur les paramètres physico-chimiques de la mare pédagogique du CIV et notamment sur les variations de température de …
Une application pour simuler la cinétique de l’hydrolyse de l’amidon
par Fabrice PELLEGRIN Le logiciel « Hydrolyse de l’amidon », écrit en HTML5, est utilisable sur tous les supports. Il permet de simuler la réalisation d’expériences sur la catalyse enzymatique et chimique de l’amidon en faisant varier les conditions de pH et de température.
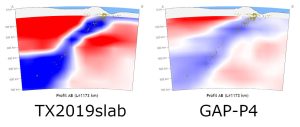
De nouveaux modèles de tomographie sismique dans Tectoglob3D
par Philippe Cosentino La tomographie sismique permet, à partir des écarts de vitesse de propagation des ondes sismiques, de mettre en évidence la présence d’anomalies thermiques dans le manteau. Depuis mars 2024, Tectoglob3D propose un nouveau modèle de tomographie qui donne des résultats exploitables aussi bien dans des contextes de convergence et de divergence. Cet …
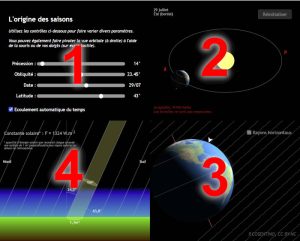
Etudier l’inégale répartition de l’énergie solaire à l’aide de l’application « Origine des saisons »
par Philippe COSENTINO Le programme d’enseignement scientifique (2023)de la classe de première aborde en détail la question de la répartition (dans l’espace et dans le temps) de l’énergie solaire sur Terre. L’application « Origine des saisons » permet de faire varier différents paramètres astronomiques et d’en étudier les effets sur la réception de l’énergie solaire par notre …