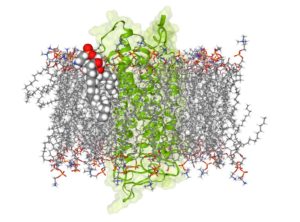
par Philippe Cosentino Dans le programme d’enseignement scientifique de la classe de première, la membrane plasmique fait partie des notions exigibles. Extrait du B.O. : « La cellule est un espace séparé de l’extérieur par une membrane plasmique. Cette membrane est constituée d’une bicouche lipidique et de protéines. » Il est tout à fait possible d’utiliser l’application …
Catégorie : Modèles moléculaires
Imprimer une molécule en 3D à l’aide du logiciel Libmol
par Philippe Cosentino Cette courte capsule vidéo (moins de 2 minutes) vous expliquera comment, en quelques clics de souris, et sans aucun savoir-faire technique particulier, imprimer en 3D une molécule à partir du logiciel Libmol. https://acamedia.ac-nice.fr/edit_video.php?vid=6321 Liens Lien vers la vidéo sur Acamédia Lien vers le logiciel Libmol
Un modèle moléculaire simplifié pour étudier la complémentarité lactase-lactose
Par Philippe Cosentino La lactase (bêta galactosidase) est un tétramère, ce qui rend complexe son étude en classe. Modèle original : 1JYN (PDB) Nous proposons un modèle simplifié, limité à un seul monomère, et débarrassé de toutes les molécules de solvant, les ions, l’eau (plusieurs centaines) présentes initialement dans le fichier (H2O, DMS, Na+, Mg++, …
Imprimante 3D et modèles moléculaires
Les imprimantes 3D permettent de réaliser des modèles en matière plastique. Il est relativement facile de réaliser un modèle moléculaire dans cette matière à partir d’un fichier PDB. Ce document relate une expérimentation d’impression 3D de modèles moléculaires. Il a été rédigé en 2013, depuis de nouvelles possibilités sont apparues dans ce domaine, notamment avec …
Comparaison de modèles moléculaires d’ADN et d’ARN
Comparaison interactive de deux modèles moléculaires correspondant à un fragment d’ADN et d’ARN. Version en ligne : https://www.pedagogie.ac-nice.fr/svt/productions/molecules/adnarnmol/ Auteur : Philippe COSENTINO
Modèles moléculaires pour l’étude de la mucoviscidose
Comparaison de modèles complets ou simplifiés de la molécule de CFTR, entre un individu normal ou frappé de mucoviscidose. https://www.pedagogie.ac-nice.fr/svt/productions/molecules/mucoviscidose/ Auteur : Philippe Cosentino
Comparaison de modèles moléculaires et de mailles cristallines (matière issue du vivant versus matière minérale)
Cette application permet d’afficher diverses molécules et mailles cristallines, afin de compter les atomes qui les constituent. https://www.pedagogie.ac-nice.fr/svt/productions/molecules/vivant-inerte/ Autre version (davantage de molécules) : https://www.pedagogie.ac-nice.fr/svt/productions/molecules/vivant-inerte/index2.htm Auteur : Philippe Cosentino
Modèle moléculaire d’endonucléase et réparation de l’ADN
Modèle moléculaire permettant d’étudier le complexe formé par une endonucléase et un fragment d’ADN présentant un dimère de thymine. https://www.pedagogie.ac-nice.fr/svt/productions/molecules/enzyme-reparation/ Auteur : Philippe Cosentino
Modèle moléculaire de la chaîne bêta de l’hémoglobine normale / drépanocytaire
Comparaison de deux modèles moléculaires de chaîne bêta d’hémoglobine, dans le cas de la drépanocytose. https://www.pedagogie.ac-nice.fr/svt/productions/molecules/drepamol/ Auteur : Philippe Cosentino
Activités autour de l’anticorps anti GP-120
Application permettant de réaliser diverses opérations sur l’anticorps anti GP-120 : colorations, mise en évidence d’acides aminés précis … En mettant en évidence les acides-aminés variables (à partir d’un document fourni par l’enseignant), l’élève pourra ainsi mettre en évidence la partie impliquée dans la reconnaissance de l’antigène. https://www.pedagogie.ac-nice.fr/svt/productions/molecules/anticorps/ Auteur : Philippe Cosentino
- 1
- 2