Par Sébastien Gruszka
Objectifs :
Ce projet a été réalisé dans le cadre des Traams 2019 de l’académie de Nice « Modélisation et codage en SVT ».
L’objectif est de faire un lien entre les enseignements d’ICN (ou technologie en collège) et de SVT, en faisant coder (en scratch) aux élèves d’ICN un programme modélisant le métabolisme des levures de l’équipement spécialisé dont les enzymes. Il est possible de prolonger cette activité en illustrant le contrôle du métabolisme par des conditions de milieu comme la température (notions abordées dans l’ancien programme de SVT ).
Les élèves doivent élaborer un programme qui modélise les réactions métaboliques qui se produisent dans le bio réacteur afin d’aider la compréhension des résultats expérimentaux obtenus par EXAO.
Scratch , logiciel connu des élèves depuis le collège, présente l’avantage de programmer sans avoir de problème de syntaxe, tout en permettant d’aborder les notions de conditions, boucles, variables essentielles à l’élaboration du projet.
Présentation du programme :
- L’écran d’accueil présente les différents « agents » utilisés dans le modèle O2, CO2, , éthanol, glucose et levure qui sont représentés par des boules de différentes couleurs.
- Début du programme (en cliquant sur le bioréacteur) : choix des différents paramètres pour rendre le modèle interactif et modifiable (quantité de molécule, levure mutée et éventuellement effet de la température ).
- Le programme se lance et on observe la levure, l’O2 et le CO2 qui se déplacent : pas de réaction si levures sans glucose.
- Ajout de glucose en cliquant sur la seringue.
- Si la levure touche O2 (jaune) et glucose (vert) elle produit du CO2 (violet)
et l’O2 et le glucose disparaissent = respiration. - Quand la levure n’a pas respiré depuis 10 secondes (il ne reste plus beaucoup d’O2) la levure commence la fermentation = si la levure touche glucose (vert)
elle produit du CO2 (violet) et de l’éthanol (noir). - Afin de vérifier que le modèle est en adéquation avec les résultats obtenus par EXAO, on peut récupérer l’évolution des paramètres mesurés qui sont enregistrés dans des listes afin de tracer un graphique à l’aide d’un tableur (voir vidéo).
- Si la levure sélectionnée est mutée, elle ne consomme pas l’O2 donc elle ne réalise que la fermentation : Le métabolisme dépend de l’équipement spécialisé de chaque cellule (organites, macromolécules dont les enzymes).
- Si la température sélectionnée est inférieure à 20°C, la vitesse de déplacement des lutins est plus faible donc les réactions sont plus lentes : influence du l’environnement sur le métabolisme (ancien programme).
Résultats obtenus
Ci-dessous, présentation vidéo du projet :
https://acamedia.ac-nice.fr/watch_video.php?v=SSS8D1U87A9B
Proposition pédagogique : Faire coder le programme aux élèves avec scratch
- Présentation du projet en utilisant le padlet (sans les consignes)
- Avant de commencer à programmer, donner le projet à compléter ainsi que quelques questions préparant la programmation.
- Au cours des séances suivantes les élèves codent pas à pas (ajout des consignes au fur et à mesure sur le padlet).
- Les notions de programmation indispensables pour réaliser ce projet et qui doivent avoir été abordées avant sont :
- Variables (évolution O2, CO2, Ethanol, glucose, temps, durée de déplacement…)
- Utilisation de la fonction clonage
- Conditions (si, si sinon…)
- Communication entres lutins (messages)
- Réaliser un questionnaire
L’enseignement d’ICN sera remplacé à la rentrée 2019 par la discipline SNT qui n’a pas les même objectifs.Il sera difficile de réaliser ce projet car le langage de programmation est imposé (Python) et la place de la programmation est bien moindre.
Il sera cependant possible d’adapter la démarche dans le cadre d’un projet d’élève en NSI (première ou terminale) codé en Python (exemple) ou dans le cadre des EPI (SVT Technologie Mathématiques) en cycle 4, classe de 3ème.
Proposition pédagogique : Utiliser le programme réalisé par les élèves en TP de SVT
L’utilisation du programme peut être réalisée par d’autres élèves dans un TP de SVT mais ne remplace en rien l’étude du métabolisme des levures par EXAO. Ce programme est élaboré pour aider à la compréhension des résultats expérimentaux pas pour les remplacer.
L’utilisation de cette modélisation ne peut donc pas être placée au le début d’une démarche pédagogique.
Connaissances :
Pour assurer les besoins fonctionnels d’une cellule, de nombreuses transformations biochimiques s’y déroulent : elles constituent son métabolisme.
Une voie métabolique est une succession de réactions biochimiques transformant une molécule en une autre.
- Etape 1 : Modélisation du métabolisme des levures non mutée à 40 °C
- Passer en mode plein écran
- Cliquer sur le drapeau Vert
- Cliquer sur la légende pour comprendre la modélisation
- Cliquer sur le dispositif EXAO pour commencer
- Choisir les conditions de modélisation
- CO2 = 1
- Glucose = 100
- O2 = 50
- Température = 40°C
- Levure mutée = non
- Cliquer sur la seringue pour injecter le glucose
- Cliquer sur l’icône I pour afficher la légende si besoin
Q1 : Indiquer ce que fait la levure quand on ajoute le glucose.
Aides : observer les différentes boules, observer l’évolution du nombre de boules…
Q2 : Indiquer à partir de quel moment se forme de l’éthanol.
Q3 : Indiquer à partir de quelle(s) molécule(s) se forme l’éthanol.
-
- Sortir du mode plein écran
- Exporter les données enregistrées :
- Cliquer sur voir l’intérieur
- Cliquer sur le camembert
- Cliquer Droit sur la liste Graphique
- Cliquer sur Export
- Enregistrer le fichier .txt
- Ouvrir le fichier à l’aide de Libre Office Calc
- Tracer un graphique montrant l’évolution des différents paramètres étudiés en fonction du temps
Q4 : Décrire l’évolution des paramètres enregistrés (O2, CO2 et Ethanol) au cours de l’expérience
Q5 : Interpréter l’évolution des paramètres enregistrés (production / consommation) au cours de l’expérience
Q6 : Montrer que les levures sont capables de développer 2 types de métabolisme en fonction de leur environnement.
Connaissances :
Le métabolisme dépend de l’équipement spécialisé de chaque cellule (organites, macromolécules dont les enzymes)


- Étape 2 version 1 : Modélisation de l’influence de l’équipement spécialisé des levures sur leur métabolisme.
Les métabolismes des levures (respiration ou fermentation) se réalisent grâce à des molécules spécialisées appelées enzymes.
La levure mutante possède une enzyme différente de la levure non mutée.
-
- A l’aide du programme modéliser l’influence de cette enzyme différente sur le métabolisme de la levure.
Les élèves doivent faire varier seulement le paramètre « levure mutée » et tracer un graphique afin de le comparer avec la première modélisation.
Q7 : En comparant les graphiques obtenus grâce aux modélisations, indiquer le rôle de cette enzyme dans le métabolisme des levures.
- Étape 2 version 2 : Modélisation de l’influence de l’équipement spécialisé des levures sur leur métabolisme.
Cette 2ème version a pour objectif de faire remixer le programme (simplifié) aux élèves en liant ainsi le codage (prérequis de collège) et les SVT (on peut également envisager cette activité en Python en lien avec SNT).
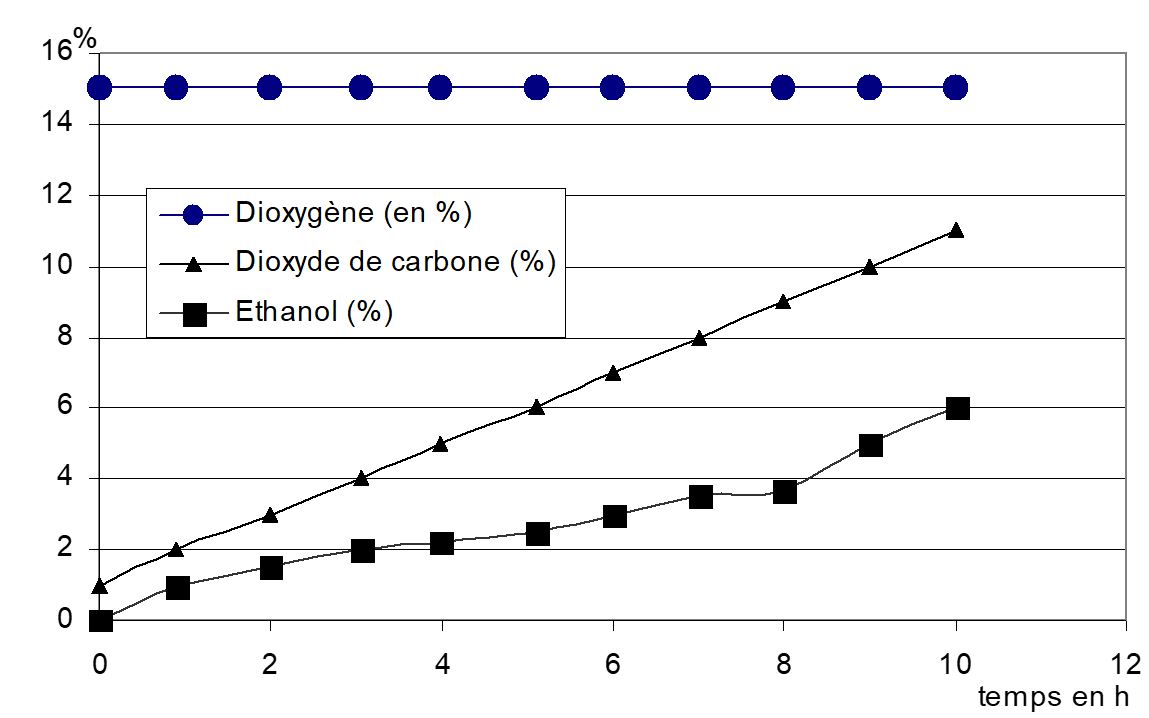
Les métabolismes des levures (respiration ou fermentation) se réalise grâce à des molécules spécialisées appelées enzymes.
La levure mutante possède une enzyme différente de la levure non mutée ce qui modifie son métabolisme :
-
-
- Remixer ce programme afin que la levure mutante est bien le métabolisme enregistré sur le graphique ci dessus.
-
Aides éventuelles :
- Lire le code et repérer comment sont coder les 2 métabolismes (respiration et fermentation).
- Supprimer les blocs qui permettent à la levure de respirer.
- Supprimer le bloc qui oblige la levure à attendre que la respiration n’ai pas eu lieu pendant 10secondes avant de fermenter.
- Solution
-
-
- A l’aide de votre programme remixé modéliser l’influence de cette enzyme différente sur le métabolisme de la levure.
-
Les élèves doivent tracer un graphique afin de le comparer avec la première modélisation.
Q8 : En comparant les graphiques obtenus grâce aux modélisations, indiquer le rôle de cette enzyme dans le métabolisme des levures.