par Philippe Cosentino
Cet article a pour objet d’expliquer comment construire des phénogrammes (arbres de similarité) avec Genigen2, comme on pouvait le faire jusqu’à présent avec Phylogène.
Introduction
Phylogène est un logiciel unique en son genre, et à l’heure où j’écris cet article il reste le seul capable de construire des cladogrammes (arbres phylogénétiques, matérialisant le degré de parenté entre des clades) à partir du partage d’apomorphies (pouce opposable, plumes etc.).
Phylogène permet également de construire des phénogrammes, c’est à dire des arbres matérialisant le degré de similarité (et non de parenté) entre des séquences.
Remarque : ces arbres sont parfois présentés comme des « arbres de parentés » ou des « arbres phylogénétiques ». Ce n’est pas tout à fait exact. Ces arbres matérialisent avant tout le degré de similarité entre les séquences. Pour les assimiler à des arbres de parenté, il faut faire l’hypothèse que le degré de similarité est proportionnel au degré de parenté (ou encore que chaque mutation a valeur d’innovation). La validité de cette hypothèse est discutable, le rythme auquel s’accumulent les mutations au cours du temps pouvant varier, ainsi que la durée des générations etc.
Les données à partir desquelles Phylogène construit ces arbres sont fournies sous la forme de fichiers .aln (aln pour « alignement »), c’est à dire de fichiers contenant des séquences déjà alignées par l’algorithme ClustalW. Certains de ces fichiers sont déjà intégrés dans les collections de Phylogène, d’autres sont fournis par l’enseignant (c’est le cas de certains sujets d’ECE).
Contrairement aux cladogrammes, que seul Phylogène permet actuellement de construire, ces mêmes phénogrammes peuvent désormais être construits avec Géniegen2. Attention toutefois, pour que cette fonctionnalité soit disponible sur la version exécutable il faut que celle-ci ait été téléchargée après le 18 janvier 2022.
Construire un arbre avec Geniegen2
Pour construire un arbre avec Geniegen2, il faut tout d’abord charger un ensemble de séquences homologues, ou du moins comparables. Elles peuvent être de nature nucléotidiques ou polypeptidiques.
Ces séquences peuvent être chargées à partir de la banque de Geniegen2 (il existe même des « Packs » de séquences optimisés pour construire des arbres), mais également sous la forme de fichiers .edi (format « Anagène »), .fasta et bien entendu .aln (le format utilisé par Phylogène).
A noter que vous pourrez récupérer ces fichiers .aln directement dans les dossiers de Phylogène.

Avec le mot clé « parenté », on trouve des « packs » qui se prêtent fort bien à la construction d’arbres.
Remarque : le format .aln n’est pas propre à Phylogène. C’est un format appelé ClustalW propre à certains logiciels tels que ClustalX, qui exportent ainsi le résultat de leurs alignements.
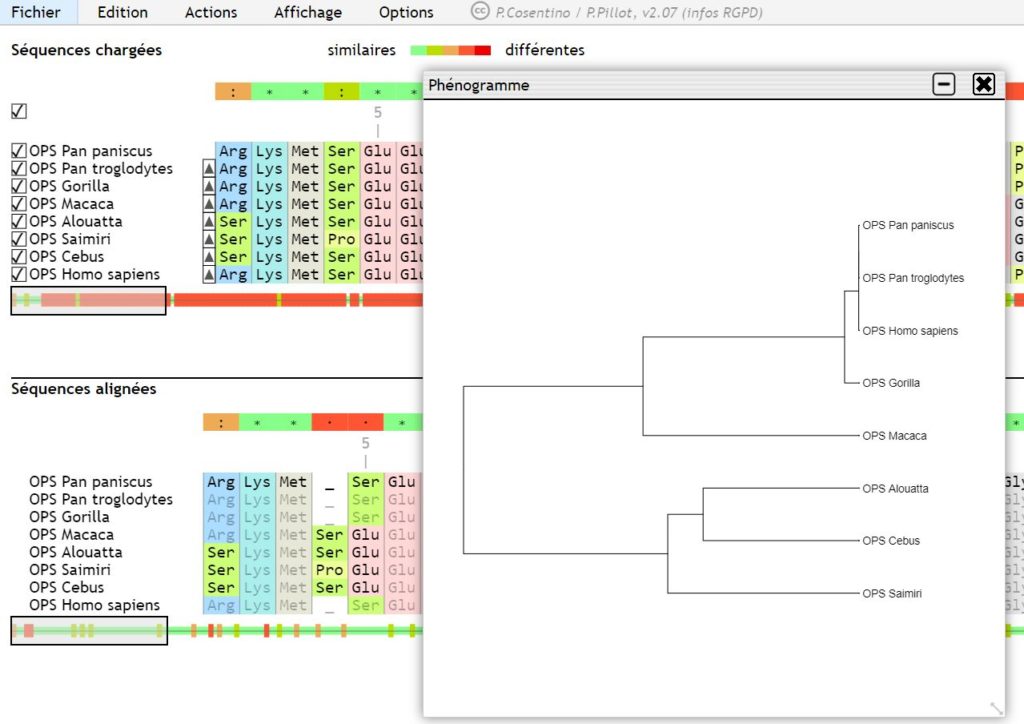
Une fois les séquences chargées, on constate qu’elles sont déjà alignées, conformément au contenu du fichier .aln.
Il ne reste plus qu’à afficher l’arbre, via le menu « Affichage ».
L’arbre apparaît alors dans une fenêtre, qu’il est possible de redimensionner.
A noter qu’il est également possible d’afficher la matrice de similarité, en passant par le menu « Affichage » puis « Tableau de comparaison ». Pour afficher plutôt la matrice de distance, il suffit de décocher la case « similarité ».
UPGMA ou WPGMA ?
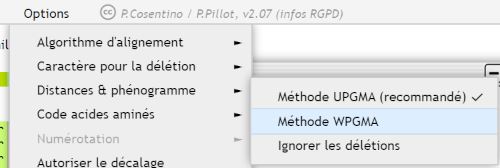
Il existe plusieurs algorithmes aboutissant à des phénogrammes légèrement différents à partir de la même matrice de distance.
Geniegen2 propose deux algorithmes différents, nommés UPGMA et WPGMA (Phylogène propose UPGMA et NJ), et vous pouvez passer de l’un à l’autre via le menu « Options ».
Les différences sont généralement minimes, mais dans certains cas on obtient des nœuds différents en changeant de méthode. Dans tous les cas il est bon de garder en tête qu’il n’existe pas UN SEUL arbre valide pour représenter les degrés de similarité entre plusieurs séquences, et qu’un algorithme peut donner de meilleurs résultats dans un cas qu’un autre dans une autre situation.
Une fonctionnalité que l’on peut cacher aux élèves
Dans le cas où l’enseignant souhaite faire construire l’arbre par les élèves, cette fonctionnalité peut devenir gênante. Pour faire disparaître l’affichage du phénogramme des menus de Geniegen2, il suffit de lui fournir un fichier .edi dont l’extension sera renommée en .nt.edi
Si par exemple la séance de travaux pratiques repose sur l’exploitation de séquences d’opsines, enregistrées dans un fichier nommé opsines.edi, il suffit de renommer ce dernier opsines.nt.edi
Une fois ce fichier ouvert par l’élève, la fonctionnalité aura irréversiblement disparu du menu de Geniegen2, jusqu’à sa fermeture.
Si les séquences ne sont pas fournies sous la forme d’un fichier, mais sont présentes dans la banque, il suffit que l’enseignant, lorsqu’il prépare sa séance, charge les séance à partir de la banque, les exporte au format .edi, et renomme le fichier obtenu.
Conclusion
Phylogène reste incontournable pour construire des cladogrammes à partir de caractères anatomiques (et autres innovations évolutives).
Cependant, pour construire des phénogrammes à partir de données moléculaires, l’alternative existe et consiste à utiliser Geniegen2.
Cette solution permettra notamment à des enseignants n’utilisant plus Phylogène au lycée de pouvoir faire construire des arbres à partir de données moléculaires à leurs élèves, notamment dans le cadre de l’évaluation des compétences expérimentales. Dans ce cas il faudra veiller à fournir le bon fichier .aln aux élèves, à ne rien modifier d’autre (que le nom du logiciel) dans le sujet, et à utiliser la version exécutable de Geniegen2 (pas d’internet).
Enfin, l’enseignant soucieux d’assurer une continuité pédagogique avec des élèves contraints de rester à leur domicile pourra facilement utiliser Geniegen2, qui fonctionne en ligne (pas d’installation) et permet même de partager directement les séquences de travail via un lien.
Liens
Article (site de l’ENS) expliquant les méthodes UPGMA et WPGMA
Article présentant Phylogène (ENS)
Activité utilisant les arbres de Geniegen2 versus Phylogène (sur l’académie de Lyon)